Metodi di ingegneria genetica per la produzione di proteine. Nuove proteine geneticamente modificate basate su anticorpi ricombinanti contro il TNF Efimov Grigory Aleksandrovich. Studio delle proprietà biologiche del sensore fluorescente Vhh41-KTNF in vitro e in vivo
Inviare il tuo buon lavoro nella knowledge base è semplice. Utilizza il modulo sottostante
Studenti, dottorandi, giovani scienziati che utilizzano la base di conoscenze nei loro studi e nel loro lavoro ti saranno molto grati.
postato su http://www.allbest.ru/
Lavoro del corso
disciplina: Biotecnologie agrarie
sul tema: “Ingegneria delle proteine”
- Saggio
- introduzione
- I. Ingegneria delle proteine
- 1.1 Il concetto di ingegneria proteica. Storia dello sviluppo
- II. Esempi di proteine ingegnerizzate
- 3.3 Alcuni risultati dell'ingegneria proteica.
- Conclusione
- Bibliografia
Argomento: Ingegneria delle proteine.
Parole chiave: biotecnologia, ingegneria genetica, proteine, codice genetico, gene, DNA, RNA, ATP, peptidi, epitopo.
Scopo del corso: studiare il concetto di “ingegneria proteica” e le potenziali possibilità del suo utilizzo.
Potenziali opportunità dell’ingegneria proteica:
1. Modificando la forza di legame della sostanza da convertire - il substrato - con l'enzima, è possibile aumentare l'efficienza catalitica complessiva della reazione enzimatica.
2. Aumentando la stabilità della proteina in un ampio intervallo di temperature e acidità, può essere utilizzata in condizioni in cui la proteina originale si denatura e perde la sua attività.
3. Creando proteine che possono funzionare in solventi anidri, è possibile effettuare reazioni catalitiche in condizioni non fisiologiche.
4. Modificando il centro catalitico di un enzima, è possibile aumentarne la specificità e ridurre il numero di reazioni collaterali indesiderate
5. Aumentando la resistenza della proteina agli enzimi che la degradano, la procedura per la sua purificazione può essere semplificata.
6. Modificando una proteina in modo che possa funzionare senza la sua consueta componente non aminoacidica (vitamina, atomo di metallo, ecc.), può essere utilizzata in alcuni processi tecnologici continui.
7. Modificando la struttura delle sezioni regolatrici dell'enzima, è possibile ridurre il grado di inibizione da parte del prodotto della reazione enzimatica in base al tipo di feedback negativo e quindi aumentare la resa del prodotto.
8. È possibile creare una proteina ibrida che abbia le funzioni di due o più proteine.
9. È possibile creare una proteina ibrida, una delle sezioni della quale facilita il rilascio della proteina ibrida dalla cellula in coltura o la sua estrazione dalla miscela.
introduzione
Da tempo immemorabile, la biotecnologia è stata utilizzata principalmente nell'industria alimentare e leggera: nella vinificazione, nella panificazione, nella fermentazione dei latticini, nella lavorazione del lino e del cuoio, basata sull'utilizzo di microrganismi. Negli ultimi decenni le possibilità della biotecnologia si sono ampliate enormemente. Ciò è dovuto al fatto che i suoi metodi sono più redditizi di quelli convenzionali per il semplice motivo che negli organismi viventi le reazioni biochimiche catalizzate da enzimi avvengono in condizioni ottimali (temperatura e pressione), sono più produttive, rispettose dell'ambiente e non richiedono sostanze chimiche. reagenti che avvelenano l'ambiente.
Gli oggetti della biotecnologia sono numerosi rappresentanti di gruppi di organismi viventi: microrganismi (virus, batteri, protozoi, lieviti), piante, animali, nonché cellule isolate da essi e componenti subcellulari (organelli) e persino enzimi. La biotecnologia si basa su processi fisiologici e biochimici che si verificano nei sistemi viventi, che determinano il rilascio di energia, la sintesi e la scomposizione dei prodotti metabolici e la formazione di componenti chimici e strutturali della cellula.
La direzione principale della biotecnologia è la produzione, utilizzando microrganismi e cellule eucariotiche in coltura, di composti biologicamente attivi (enzimi, vitamine, ormoni), farmaci (antibiotici, vaccini, sieri, anticorpi altamente specifici, ecc.), nonché composti preziosi ( additivi per mangimi, ad esempio aminoacidi essenziali, proteine dei mangimi, ecc.).
I metodi di ingegneria genetica hanno permesso di sintetizzare in quantità industriali ormoni come l'insulina e la somatotropina (ormone della crescita), necessari per il trattamento delle malattie genetiche umane.
La biotecnologia non risolve solo problemi specifici della scienza e della produzione. Ha un compito metodologico più globale: espande e accelera la portata dell'impatto umano sulla natura vivente e promuove l'adattamento dei sistemi viventi alle condizioni dell'esistenza umana, ad es. alla noosfera. La biotecnologia, quindi, agisce come un potente fattore nell’evoluzione adattativa antropogenica.
La biotecnologia, l'ingegneria genetica e quella cellulare hanno prospettive promettenti. Man mano che compaiono sempre più nuovi vettori, le persone li utilizzeranno per introdurre i geni necessari nelle cellule di piante, animali e esseri umani. Ciò consentirà di eliminare gradualmente molte malattie umane ereditarie, costringere le cellule a sintetizzare i farmaci necessari e i composti biologicamente attivi, e quindi direttamente le proteine e gli aminoacidi essenziali utilizzati negli alimenti. Utilizzando metodi già padroneggiati dalla natura, i biotecnologi sperano di ottenere idrogeno attraverso la fotosintesi, il combustibile più rispettoso dell'ambiente del futuro, l'elettricità, e di convertire l'azoto atmosferico in ammoniaca in condizioni normali.
Le proprietà fisiche e chimiche delle proteine naturali spesso non soddisfano le condizioni in cui queste proteine verranno utilizzate dagli esseri umani. È necessario un cambiamento nella sua struttura primaria, che garantirà la formazione di una proteina con una struttura spaziale diversa rispetto a prima e nuove proprietà fisico-chimiche, che le consentiranno di svolgere le funzioni inerenti alla proteina naturale in altre condizioni. L'ingegneria proteica si occupa della costruzione delle proteine.
Un altro campo di applicazione dell'ingegneria proteica è la creazione di proteine in grado di neutralizzare sostanze e microrganismi utilizzabili per attacchi chimici e biologici. Ad esempio, gli enzimi idrolasi sono in grado di neutralizzare sia i gas nervini che i pesticidi utilizzati in agricoltura. Inoltre, la produzione, la conservazione e l'utilizzo degli enzimi non sono pericolosi per l'ambiente e la salute umana.
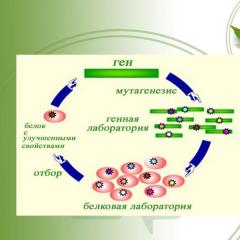
Per ottenere una proteina alterata, vengono utilizzati metodi di chimica combinatoria e viene eseguita la mutagenesi diretta, introducendo cambiamenti specifici nelle sequenze codificanti del DNA, portando a determinati cambiamenti nelle sequenze di amminoacidi. Per progettare efficacemente una proteina con le proprietà desiderate, è necessario conoscere i modelli di formazione della struttura spaziale della proteina, da cui dipendono le sue proprietà e funzioni fisico-chimiche, cioè è necessario sapere come funziona la struttura primaria della proteina , ciascuno dei suoi residui aminoacidici influenza le proprietà e le funzioni della proteina. Sfortunatamente, per la maggior parte delle proteine la struttura terziaria è sconosciuta; non sempre è noto quale amminoacido o sequenza di amminoacidi debba essere modificata per ottenere una proteina con le proprietà desiderate. Già ora gli scienziati che utilizzano l’analisi computerizzata possono prevedere le proprietà di molte proteine in base alla sequenza dei loro residui aminoacidici. Tale analisi semplificherà notevolmente la procedura per creare le proteine desiderate. Nel frattempo, per ottenere una proteina modificata con le proprietà desiderate, si procede principalmente in modo diverso: si ottengono diversi geni mutanti e si trova il prodotto proteico di uno di essi che abbia le proprietà desiderate.
Vari approcci sperimentali vengono utilizzati per la mutagenesi sito-diretta. Dopo aver ricevuto il gene modificato, questo viene integrato in un costrutto genetico e introdotto nelle cellule procariotiche o eucariotiche che sintetizzano la proteina codificata da questo costrutto genetico.
I. Ingegneria delle proteine
1.1 Il concetto di ingegneria proteica. Storia dello sviluppo
L'ingegneria proteica è una branca della biotecnologia che si occupa dello sviluppo di proteine utili o preziose. Si tratta di una disciplina relativamente nuova che si concentra sullo studio del ripiegamento delle proteine e sui principi della modifica e creazione delle proteine.
Esistono due strategie principali per l'ingegneria proteica: la modificazione diretta delle proteine e l'evoluzione diretta. Questi metodi non si escludono a vicenda; i ricercatori spesso usano entrambi. In futuro, una conoscenza più dettagliata della struttura e della funzione delle proteine, nonché i progressi nell'alta tecnologia, potrebbero espandere significativamente le possibilità dell'ingegneria proteica. Di conseguenza, anche gli aminoacidi innaturali possono essere incorporati grazie a un nuovo metodo che consente di incorporare nuovi aminoacidi nel codice genetico.
L'ingegneria delle proteine ha avuto origine all'intersezione tra la fisica e la chimica delle proteine e l'ingegneria genetica. Risolve il problema della creazione di molecole proteiche modificate o ibride con caratteristiche specifiche. Un modo naturale per eseguire tale compito è prevedere la struttura del gene che codifica per la proteina alterata, effettuarne la sintesi, la clonazione e l'espressione nelle cellule riceventi.
La prima modificazione controllata delle proteine fu effettuata a metà degli anni '60 da Koshland e Bender. Per sostituire il gruppo idrossile con un gruppo sulfidrile nel centro attivo della proteasi, la subtilisina, hanno utilizzato un metodo di modificazione chimica. Tuttavia, come si è scoperto, tale tiolsubtilisina non mantiene l'attività della proteasi.
Chimicamente, una proteina è un singolo tipo di molecola, che è una catena di poliamminoacidi o un polimero. È composto da sequenze di aminoacidi di 20 tipi. Dopo aver appreso la struttura delle proteine, le persone si sono poste la domanda: è possibile progettare sequenze di amminoacidi completamente nuove in modo che svolgano le funzioni di cui gli esseri umani hanno bisogno molto meglio delle normali proteine? Il nome Protein Engineering era appropriato per questa idea.
La gente ha iniziato a pensare a tale ingegneria negli anni '50 del 20 ° secolo. Ciò è avvenuto immediatamente dopo aver decifrato le prime sequenze di aminoacidi proteici. In molti laboratori in tutto il mondo sono stati fatti tentativi per duplicare la natura e sintetizzare chimicamente determinate sequenze di poliamminoacidi assolutamente arbitrarie.
Il chimico B. Merrifield è riuscito maggiormente in questo. Questo americano è riuscito a sviluppare un metodo estremamente efficace per la sintesi delle catene di poliamminoacidi. Per questo alla Merrifield venne assegnato il Premio Nobel per la Chimica nel 1984.
Figura 1. Schema di come funziona l'ingegneria proteica.
L'americano iniziò a sintetizzare peptidi corti, compresi gli ormoni. Allo stesso tempo, costruì un automa - un "robot chimico" - il cui compito era produrre proteine artificiali. Il robot fece scalpore negli ambienti scientifici. Tuttavia, divenne presto chiaro che i suoi prodotti non potevano competere con ciò che la natura produce.
Il robot non è riuscito a riprodurre accuratamente le sequenze di aminoacidi, ovvero ha commesso degli errori. Ha sintetizzato una catena con una sequenza e l'altra con una leggermente modificata. In una cellula, tutte le molecole di una proteina sono idealmente simili tra loro, cioè le loro sequenze sono assolutamente identiche.
C'era un altro problema. Anche le molecole che il robot ha sintetizzato correttamente non hanno assunto la forma spaziale necessaria per il funzionamento dell'enzima. Pertanto, il tentativo di sostituire la natura con i metodi consueti della chimica organica ha portato a un successo molto modesto.
Gli scienziati potevano solo imparare dalla natura, cercando le necessarie modifiche delle proteine. Il punto è che in natura avvengono costantemente mutazioni che portano a cambiamenti nelle sequenze di aminoacidi delle proteine. Se selezioni mutanti con le proprietà necessarie che elaborano un particolare substrato in modo più efficiente, puoi isolare da tale mutante un enzima alterato, grazie al quale la cellula acquisisce nuove proprietà. Ma questo processo richiede un periodo di tempo molto lungo.
Tutto è cambiato quando è apparsa l’ingegneria genetica. Grazie a lei, hanno iniziato a creare geni artificiali con qualsiasi sequenza nucleotidica. Questi geni sono stati inseriti in molecole vettoriali preparate e il DNA è stato introdotto in batteri o lieviti. Lì, una copia dell'RNA è stata prelevata dal gene artificiale. Di conseguenza, è stata prodotta la proteina richiesta. Sono stati esclusi errori nella sua sintesi. La cosa principale era selezionare la giusta sequenza di DNA, dopodiché il sistema enzimatico della cellula avrebbe svolto il suo lavoro in modo impeccabile. Possiamo quindi concludere che l’ingegneria genetica ha aperto la strada all’ingegneria proteica nella sua forma più radicale.
1.2 Strategie di ingegneria proteica
Modificazione proteica mirata. Nella modifica mirata delle proteine, lo scienziato utilizza la conoscenza dettagliata della struttura e della funzione della proteina per apportare le modifiche desiderate. In generale, questo metodo ha il vantaggio di essere poco costoso e tecnicamente semplice, poiché la tecnica della mutagenesi sito-diretta è ben sviluppata. Tuttavia, il suo principale svantaggio è che spesso mancano informazioni sulla struttura dettagliata di una proteina e, anche quando la struttura è nota, può essere molto difficile prevedere l'effetto delle varie mutazioni.
Gli algoritmi del software di modificazione delle proteine si sforzano di identificare nuove sequenze di amminoacidi che richiedono poca energia per formare una struttura bersaglio predefinita. Sebbene la sequenza da trovare sia ampia, il requisito più difficile per la modificazione delle proteine è un modo veloce, ma preciso, per identificare e definire la sequenza ottimale, in contrapposizione a sequenze subottimali simili.
Evoluzione diretta. Nell'evoluzione diretta, la mutagenesi casuale viene applicata a una proteina e viene effettuata la selezione per selezionare varianti che hanno determinate qualità. Successivamente, vengono applicati più cicli di mutazione e selezione. Questo metodo imita l'evoluzione naturale e generalmente produce risultati superiori per la modifica diretta.
Un'ulteriore tecnica nota come mescolamento del DNA mescola e identifica parti di varianti riuscite per produrre risultati migliori. Questo processo imita le ricombinazioni che avvengono naturalmente durante la riproduzione sessuale. Il vantaggio dell'evoluzione diretta è che non richiede una conoscenza preliminare della struttura delle proteine, né è necessario poter prevedere quale effetto avrà una determinata mutazione. In effetti, i risultati degli esperimenti sull’evoluzione diretta sono sorprendenti perché i cambiamenti desiderati sono spesso causati da mutazioni che non dovrebbero avere tale effetto. Lo svantaggio è che questo metodo richiede un rendimento elevato, cosa non possibile per tutte le proteine. Grandi quantità di DNA ricombinante devono essere mutate e i prodotti devono essere selezionati per la qualità desiderata. L’enorme numero di opzioni spesso richiede l’acquisto di robotica per automatizzare il processo. Inoltre, non è sempre facile individuare tutte le qualità di interesse.
II. Esempi di proteine ingegnerizzate
L'ingegneria proteica può basarsi sulla modificazione chimica di una proteina finita o su metodi di ingegneria genetica che consentono di ottenere versioni modificate di proteine naturali.
La progettazione di uno specifico catalizzatore biologico viene effettuata tenendo conto sia della specificità della proteina che dell'attività catalitica del complesso organometallico. Ecco alcuni esempi di tali modifiche effettuate per ottenere “complessi bioorganici semisintetici”. La mioglobina del capodoglio è in grado di legare l'ossigeno, ma non ha attività biocatalitica. Come risultato della combinazione di questa biomolecola con tre complessi di trasferimento di elettroni contenenti rutenio, che si legano ai residui di istidina sulla superficie delle molecole proteiche, si forma un complesso in grado di ridurre l'ossigeno e contemporaneamente ossidare numerosi substrati organici, come come ascorbato, ad una velocità quasi identica a quella dell’ascorbato ossidasi naturale. In linea di principio, le proteine possono essere modificate in altri modi. Consideriamo ad esempio la papaina. È uno degli enzimi proteolitici ben studiati per i quali è stata determinata una struttura tridimensionale. Vicino al residuo di cisteina-25 sulla superficie della molecola proteica è presente un solco esteso in cui avviene la reazione di proteolisi. Questo sito può essere alchilato da un derivato della flavina senza modificare l'accessibilità del potenziale sito di legame del substrato. Tali flavopapaine modificate sono state utilizzate per l'ossidazione delle M-alchil-1,4-diidronicotinammidi e l'attività catalitica di alcune di queste proteine modificate era significativamente superiore a quella delle flavoproteine-NADH deidrogenasi naturali. Pertanto, è stato possibile creare un enzima semisintetico molto efficace. L'uso di flavine con sostituenti posizionati altamente attivi che attraggono gli elettroni può rendere possibile lo sviluppo di catalizzatori efficaci per la riduzione della nicotina ammide.
Gli importanti progressi conseguiti di recente nella sintesi chimica del DNA hanno aperto opportunità fondamentalmente nuove per l’ingegneria proteica: la progettazione di proteine uniche che non si trovano in natura. Ciò richiede un ulteriore sviluppo della tecnologia, in modo che la modifica dei geni utilizzando metodi di ingegneria genetica porti a cambiamenti prevedibili nelle proteine, a un miglioramento delle loro caratteristiche funzionali ben definite: numero di turnover, Km per un substrato specifico, stabilità termica, temperatura ottimale, stabilità e attività in solventi non acquosi, specificità del substrato e della reazione, fabbisogno di cofattori, pH ottimale, resistenza alle proteasi, regolazione allosterica, peso molecolare e struttura delle subunità. Tipicamente, tale miglioramento è stato ottenuto attraverso la mutagenesi e la selezione e, più recentemente, attraverso la modificazione chimica e l'immobilizzazione. Per progettare con successo un tipo specifico di molecola proteica, è necessario identificare una serie di modelli fondamentali che collegano le caratteristiche strutturali delle proteine e le loro proprietà desiderate. Pertanto, conoscendo l'esatta struttura cristallina della molecola proteica in studio, è possibile individuare quelle parti di essa che andrebbero specificatamente modificate per aumentarne l'attività catalitica. Tale modifica può consistere nel cambiare la sequenza aminoacidica della proteina.
Un altro esempio è l'implementazione della mutagenesi sito-specifica. Succede come segue. Il gene della proteina che interessa al ricercatore viene clonato e inserito in un portatore genetico adatto. Successivamente viene sintetizzato un primer oligonucleotidico con la mutazione desiderata, la cui sequenza, composta da dieci a quindici nucleotidi, è sufficientemente omologa ad una determinata regione del gene naturale ed è quindi in grado di formare con esso una struttura ibrida. Questo primer sintetico viene utilizzato dalle polimerasi per avviare la sintesi di una copia complementare del vettore, che viene poi separata dall'originale e utilizzata per la sintesi controllata della proteina mutante. Un approccio alternativo si basa sulla scissione della catena, sulla rimozione del sito da modificare e sulla sua sostituzione con un analogo sintetico con la sequenza nucleotidica desiderata.
La tirosil-tRNA sintetasi catalizza la reazione di aminoacilazione del tRNA della tirosina, che comporta l'attivazione della tirosina da parte dell'ATP per formare tirosil adenilato. Il gene per questo enzima, isolato dal Bacillus stearothermophilus, è stato inserito nel batteriofago M13. Le proprietà catalitiche dell'enzima, in particolare la sua capacità di legarsi al substrato, sono state poi alterate mediante modifiche sito-specifiche. Pertanto, la treonina-51 è stata sostituita dall'alanina. Ciò ha comportato un duplice aumento del legame con il substrato, apparentemente a causa dell'incapacità di formare un legame idrogeno tra questo residuo e il tirosil adenilato. Quando si sostituisce l'alanina con la prolina, la configurazione della molecola enzimatica viene interrotta, ma la capacità di legare il substrato aumenta di cento volte, poiché la sua interazione con l'istidina-48 è facilitata. Simili cambiamenti sito-specifici sono stati ottenuti nella β-lattamasi e di solito sono accompagnati dall'inattivazione dell'enzima. La sostituzione della serina-70 con la cisteina porta alla formazione della p-tiolo lattamasi, la cui costante di legame non differisce da quella dell'enzima naturale, ma l'attività verso la penicillina è solo dell'1-2%. Tuttavia, l'attività di questo enzima mutante contro alcune cefalosporine attivate non è inferiore all'attività originale o addirittura la supera; queste proteine sono anche più resistenti alle proteasi.
Le mutazioni sito-specifiche vengono ora utilizzate per testare l'adeguatezza degli studi strutturali. In alcuni casi, sono riusciti a dimostrare che la stabilità strutturale di una proteina e la sua attività catalitica possono essere disaccoppiate. Si è accumulata una quantità sufficiente di informazioni sulla relazione tra la stabilità della struttura proteica e la sua funzione; potremmo essere in grado di mettere a punto l'attività dei catalizzatori biologici e crearne analoghi completamente sintetici. Recentemente è apparso un lavoro che riportava la clonazione del primo gene dell'enzima sintetico che codifica il frammento attivo della molecola di ribonucleasi.
III. Applicazioni dell'ingegneria proteica
La tecnologia dell'ingegneria proteica viene utilizzata (spesso in combinazione con il metodo del DNA ricombinante) per migliorare le proprietà delle proteine esistenti (enzimi, anticorpi, recettori cellulari) e creare nuove proteine che non esistono in natura. Tali proteine vengono utilizzate per creare medicinali, nella lavorazione degli alimenti e nella produzione industriale.
Attualmente, l’applicazione più popolare dell’ingegneria proteica è quella di modificare le proprietà catalitiche degli enzimi per sviluppare processi industriali “rispettosi dell’ambiente”. Da un punto di vista ambientale, gli enzimi sono i più accettabili tra tutti i catalizzatori utilizzati nell'industria. Ciò è garantito dalla capacità dei biocatalizzatori di dissolversi in acqua e di funzionare pienamente in un ambiente con pH neutro e a temperature relativamente basse. Inoltre, grazie alla loro elevata specificità, l'uso dei biocatalizzatori produce pochissimi sottoprodotti di produzione indesiderati. I processi industriali rispettosi dell'ambiente e a risparmio energetico che utilizzano biocatalizzatori sono stati da tempo introdotti attivamente nei settori chimico, tessile, farmaceutico, della pasta di legno e della carta, alimentare, energetico e in altri settori dell'industria moderna.
Tuttavia, alcune caratteristiche dei biocatalizzatori ne rendono inaccettabile l’uso in alcuni casi. Ad esempio, la maggior parte degli enzimi si degrada quando la temperatura aumenta. Gli scienziati stanno cercando di superare tali ostacoli e di aumentare la stabilità degli enzimi in condizioni di produzione difficili utilizzando tecniche di ingegneria proteica.
Oltre alle applicazioni industriali, l’ingegneria proteica ha trovato un posto degno negli sviluppi medici. I ricercatori sintetizzano proteine che possono legarsi e neutralizzare virus e geni mutanti che causano tumori; creando vaccini altamente efficaci e studiando le proteine recettoriali sulla superficie cellulare, che spesso sono bersagli per i prodotti farmaceutici. Gli scienziati alimentari utilizzano l'ingegneria proteica per migliorare le proprietà di conservazione delle proteine vegetali e degli agenti gelificanti o addensanti.
3.1 Librerie di peptidi ed epitopi
In un organismo vivente, la maggior parte dei processi biologici sono controllati attraverso specifiche interazioni proteina-proteina o proteina-acido nucleico. Tali processi includono, ad esempio, la regolazione della trascrizione genetica sotto l'influenza di vari fattori proteici, l'interazione dei ligandi proteici con i recettori sulla superficie delle cellule, nonché il legame specifico degli antigeni con gli anticorpi corrispondenti. Comprendere i meccanismi molecolari di interazione dei ligandi proteici con i recettori è di grande importanza fondamentale e applicata. In particolare, lo sviluppo di nuovi farmaci proteici inizia solitamente con l'identificazione della sequenza aminoacidica iniziale dotata dell'attività biologica richiesta (la cosiddetta sequenza “lead”). Tuttavia, i peptidi con una sequenza aminoacidica basica possono anche avere proprietà biologiche indesiderabili: bassa attività, tossicità, bassa stabilità nel corpo, ecc.
Prima dell'avvento delle librerie peptidiche, il miglioramento delle loro proprietà biologiche veniva effettuato mediante sintesi sequenziale di un gran numero di analoghi e test della loro attività biologica, il che richiedeva molto tempo e denaro. Negli ultimi anni è diventato possibile creare migliaia di peptidi diversi in breve tempo utilizzando sintetizzatori automatici. I metodi sviluppati di mutagenesi mirata hanno inoltre consentito di espandere notevolmente il numero di proteine ottenute simultaneamente e successivamente testate per l'attività biologica. Tuttavia, solo gli approcci sviluppati di recente per la creazione di librerie di peptidi hanno prodotto i milioni di sequenze di amminoacidi necessarie per individuare efficacemente i peptidi che meglio soddisfano i criteri. Tali librerie vengono utilizzate per studiare l'interazione degli anticorpi con gli antigeni, ottenere nuovi inibitori enzimatici e agenti antimicrobici, progettare molecole con l'attività biologica desiderata o conferire nuove proprietà alle proteine, come gli anticorpi.
In base ai metodi di preparazione, le librerie peptidiche sono divise in tre gruppi. Il primo gruppo comprende librerie ottenute mediante sintesi chimica di peptidi, in cui i singoli peptidi sono immobilizzati su microcarrier. Con questo approccio, dopo l'aggiunta di successivi amminoacidi nelle singole miscele di reazione ai peptidi immobilizzati su microcarrier, il contenuto di tutte le miscele di reazione viene combinato e diviso in nuove porzioni, che vengono utilizzate nella fase successiva di aggiunta di nuovi residui amminoacidici. Dopo una serie di tali passaggi, vengono sintetizzati peptidi contenenti le sequenze di amminoacidi utilizzate nella sintesi in tutti i tipi di combinazioni casuali.
Le librerie di peptidi immobilizzati su microcarrier presentano uno svantaggio significativo: richiedono l'uso di recettori purificati in forma solubile durante lo screening. Allo stesso tempo, nella maggior parte dei casi, i recettori associati alla membrana vengono spesso utilizzati nei test biologici effettuati per la ricerca di base e farmacologica. Secondo il secondo metodo, le librerie peptidiche vengono ottenute utilizzando la sintesi peptidica in fase solida, in cui in ogni fase dell'aggiunta chimica dell'amminoacido successivo alle catene peptidiche in crescita, vengono utilizzate miscele equimolari di tutti o alcuni amminoacidi precursori. Nella fase finale della sintesi, i peptidi vengono separati dal trasportatore, vale a dire trasformandoli in forma solubile. Il terzo approccio alla costruzione delle librerie peptidiche, che ora descriviamo, è diventato fattibile proprio grazie allo sviluppo di metodi di ingegneria genetica. Illustra perfettamente le capacità di tali metodi e rappresenta senza dubbio un importante progresso nella loro applicazione. A questo proposito, considereremo più in dettaglio i risultati dell'utilizzo delle librerie peptidiche nello studio degli epitopi (determinanti antigenici) delle proteine.
La tecnologia dell'ingegneria genetica per la produzione di proteine ibride ha reso possibile lo sviluppo di un metodo efficace per la produzione di peptidi corti per l'analisi della loro attività biologica. Come nel caso delle librerie genetiche, le librerie peptidiche ottenute mediante metodi di ingegneria genetica rappresentano un ampio (spesso esaustivo) insieme di peptidi corti. Due recenti osservazioni permettono di considerare una libreria di peptidi simultaneamente e come una libreria di epitopi proteici. Innanzitutto, i peptidi corti possono includere tutti i residui di amminoacidi essenziali che svolgono un ruolo importante nell’interazione degli anticorpi e sono in grado di imitare grandi determinanti antigenici delle proteine. In secondo luogo, nella maggior parte dei casi, i legami non covalenti formati tra i pochi residui amminoacidici più importanti dei ligandi proteici e i loro recettori forniscono un contributo importante all’energia totale dell’interazione ligando-recettore. Tenendo presente questo, qualsiasi peptide può essere considerato un potenziale ligando, aptene o parte del determinante antigenico di polipeptidi più grandi, e qualsiasi libreria peptidica può essere considerata una libreria di epitopi proteici o potenziali ligandi per i corrispondenti recettori proteici.
La libreria peptidica ottenuta come risultato dell'implementazione del terzo approccio, nella sua forma moderna, è un insieme di decine o addirittura centinaia di milioni di brevi sequenze di amminoacidi diverse che sono espresse sulla superficie dei virioni del batteriofago come parte della propria proteine strutturali. Ciò diventa possibile grazie all'introduzione di geni ibridi ricombinanti che codificano per proteine strutturali alterate dei suoi virioni nel genoma dei batteriofagi utilizzando metodi di ingegneria genetica. (Questo metodo è noto come phage display.) Come risultato dell'espressione di tali geni, si formano proteine ibride, ai terminali N o C delle quali sono presenti ulteriori sequenze di amminoacidi.
Biblioteche di peptidi ed epitopi troveranno il loro utilizzo anche negli studi sui meccanismi della risposta immunitaria umorale e sulle malattie del sistema immunitario. In particolare, la maggior parte delle malattie autoimmuni sono accompagnate dalla formazione di autoanticorpi contro antigeni propri dell'organismo. Questi anticorpi in molti casi servono come marcatori specifici di una particolare malattia autoimmune. Utilizzando una libreria di epitopi, in linea di principio, è possibile ottenere marcatori peptidici, con l'aiuto dei quali sarebbe possibile monitorare la specificità degli autoanticorpi durante lo sviluppo di un processo patologico sia in un singolo organismo che in un gruppo di pazienti e, inoltre, per determinare la specificità degli autoanticorpi in malattie ad eziologia sconosciuta.
Le librerie di peptidi ed epitopi possono anche essere potenzialmente utilizzate per lo screening di sieri immunitari per identificare i peptidi che interagiscono specificamente con gli anticorpi protettivi. Tali peptidi imiteranno i determinanti antigenici degli organismi patogeni e fungeranno da bersagli per gli anticorpi protettivi del corpo. Ciò consentirà l'uso di tali peptidi per la vaccinazione di pazienti privi di anticorpi contro i corrispondenti agenti patogeni. Lo studio degli epitopi utilizzando librerie peptidiche è un caso speciale di una delle tante aree del loro utilizzo negli studi applicati e fondamentali dell'interazione tra ligandi e recettori. Un ulteriore miglioramento di questo approccio dovrebbe facilitare la creazione di nuovi farmaci basati su peptidi corti e essere utile negli studi fondamentali sui meccanismi delle interazioni proteina-proteina.
3.2 Proteine reporter nelle proteine di fusione
In un altro caso, le proteine di fusione vengono utilizzate per ottenere elevati livelli di espressione di peptidi corti nelle cellule batteriche grazie alla stabilizzazione di questi peptidi all'interno delle proteine di fusione. Le proteine ibride vengono spesso utilizzate per identificare e purificare proteine ricombinanti difficili da rilevare. Ad esempio, attaccando la galattosidasi al C-terminale della proteina in studio come proteina reporter, è possibile purificare la proteina ricombinante in base all'attività della galattosidasi, determinandone i determinanti antigenici mediante metodi immunochimici. Combinando frammenti di DNA contenenti open reading frames (ORF) con i geni delle proteine reporter, è possibile purificare tali proteine ibride in base all'attività della proteina reporter e utilizzarle per immunizzare animali da laboratorio. Gli anticorpi risultanti vengono quindi utilizzati per purificare la proteina nativa, che include il polipeptide ricombinante codificato dall'ORF, e quindi identificare il frammento del gene clonato.
Con l'aiuto di proteine ibride, viene risolto anche il problema inverso della clonazione di un gene sconosciuto, al prodotto proteico di cui esistono anticorpi. In questo caso, una libreria di cloni di sequenze nucleotidiche che rappresentano ORF di geni sconosciuti viene costruita in vettori che consentono di collegare l'ORF da clonare nello stesso frame di lettura con il gene reporter. Le proteine ibride formate come risultato dell'espressione di questi geni ricombinanti vengono identificate utilizzando anticorpi utilizzando metodi di dosaggio immunoenzimatico. I geni ibridi che combinano proteine secrete e proteine reporter rendono possibile studiare in nuovi modi i meccanismi di secrezione, nonché la localizzazione e il movimento delle proteine secrete nei tessuti.
3.3 Alcuni risultati dell'ingegneria proteica
1. Sostituendo diversi residui amminoacidici del lisozima del batteriofago T4 con cisteina, è stato ottenuto un enzima con un gran numero di legami disolfuro, grazie al quale questo enzima ha mantenuto la sua attività a una temperatura più elevata.
2. La sostituzione del residuo di cisteina con un residuo di serina nella molecola dell'interferone β umano sintetizzato dall'Escherichia coli ha impedito la formazione di complessi intermolecolari, che hanno ridotto l'attività antivirale di questo farmaco di circa 10 volte.
3. La sostituzione del residuo di treonina con un residuo di prolina nella molecola dell'enzima tirosil-tRNA sintetasi ha aumentato di dieci volte l'attività catalitica di questo enzima: ha iniziato ad attaccare rapidamente la tirosina al tRNA che trasferisce questo amminoacido al ribosoma durante la traduzione.
4. Le subtilisine sono enzimi ricchi di serina che scompongono le proteine. Sono secreti da molti batteri e sono ampiamente utilizzati dagli esseri umani per la biodegradazione. Legano saldamente gli atomi di calcio, aumentandone la stabilità. Tuttavia, nei processi industriali sono presenti composti chimici che legano il calcio, dopodiché le subtilisine perdono la loro attività. Modificando il gene, gli scienziati hanno rimosso dall'enzima gli amminoacidi coinvolti nel legame del calcio e hanno sostituito un amminoacido con un altro per aumentare la stabilità della subtilisina. L'enzima modificato si è rivelato stabile e funzionalmente attivo in condizioni prossime a quelle industriali.
5. È stata dimostrata la possibilità di creare un enzima che funzioni come un enzima di restrizione che scinde il DNA in punti rigorosamente definiti. Gli scienziati hanno creato una proteina ibrida, un frammento della quale riconosceva una sequenza specifica di residui nucleotidici in una molecola di DNA, mentre l'altro DNA frammentato in questa regione.
6. L'attivatore tissutale del plasminogeno è un enzima utilizzato clinicamente per sciogliere i coaguli di sangue. Purtroppo viene rapidamente eliminato dal sistema circolatorio e deve essere somministrato ripetutamente o in dosi elevate, il che porta a effetti collaterali. Introducendo tre mutazioni mirate nel gene di questo enzima, abbiamo ottenuto un enzima a lunga vita con maggiore affinità per la fibrina degradata e con la stessa attività fibrinolitica dell'enzima originale.
7. Sostituendo un amminoacido nella molecola di insulina, gli scienziati hanno assicurato che quando questo ormone veniva somministrato per via sottocutanea a pazienti con diabete, il cambiamento nella concentrazione di questo ormone nel sangue era vicino a quello fisiologico che si verifica dopo aver mangiato.
8. Esistono tre classi di interferoni che hanno attività antivirale e antitumorale, ma presentano specificità diverse. C'era la tentazione di creare un interferone ibrido che avesse le proprietà dei tre tipi di interferoni. Sono stati creati geni ibridi che includevano frammenti di geni naturali dell'interferone di diversi tipi. Alcuni di questi geni, essendo integrati nelle cellule batteriche, hanno assicurato la sintesi di interferoni ibridi con maggiore attività antitumorale rispetto alle molecole madri.
9. L'ormone della crescita umano naturale si lega non solo al recettore di questo ormone, ma anche al recettore di un altro ormone: la prolattina. Per evitare effetti collaterali indesiderati durante il trattamento, gli scienziati hanno deciso di eliminare la possibilità che l'ormone della crescita si leghi al recettore della prolattina. Hanno raggiunto questo obiettivo sostituendo alcuni aminoacidi nella struttura primaria dell’ormone della crescita utilizzando l’ingegneria genetica.
10. Durante lo sviluppo di farmaci contro l'infezione da HIV, gli scienziati hanno ottenuto una proteina ibrida, un frammento della quale assicurava il legame specifico di questa proteina solo ai linfociti colpiti dal virus, un altro frammento effettuava la penetrazione della proteina ibrida nella cellula colpita, e un altro frammento ha interrotto la sintesi proteica nella cellula colpita, portandola alla morte.
Le proteine sono i principali bersagli dei farmaci. Attualmente sono noti circa 500 bersagli per l'azione dei farmaci. Nei prossimi anni il loro numero aumenterà fino a 10.000, il che consentirà di creare farmaci nuovi, più efficaci e sicuri. Recentemente sono stati sviluppati approcci fondamentalmente nuovi alla scoperta di farmaci: non le singole proteine, ma i loro complessi, le interazioni proteina-proteina e il ripiegamento delle proteine sono considerati come bersagli.
Conclusione
La tecnologia dell'ingegneria proteica viene utilizzata (spesso in combinazione con il metodo del DNA ricombinante) per migliorare le proprietà delle proteine esistenti (enzimi, anticorpi, recettori cellulari) e creare nuove proteine che non esistono in natura. Tali proteine vengono utilizzate per creare medicinali, nella lavorazione degli alimenti e nella produzione industriale.
Attualmente, l’applicazione più popolare dell’ingegneria proteica è quella di modificare le proprietà catalitiche degli enzimi per sviluppare processi industriali “rispettosi dell’ambiente”. Da un punto di vista ambientale, gli enzimi sono i più accettabili tra tutti i catalizzatori utilizzati nell'industria. Ciò è garantito dalla capacità dei biocatalizzatori di dissolversi in acqua e di funzionare pienamente in un ambiente con pH neutro e a temperature relativamente basse. Inoltre, grazie alla loro elevata specificità, l'uso dei biocatalizzatori produce pochissimi sottoprodotti di produzione indesiderati. I processi industriali rispettosi dell'ambiente e a risparmio energetico che utilizzano biocatalizzatori sono stati da tempo introdotti attivamente nei settori chimico, tessile, farmaceutico, della pasta di legno e della carta, alimentare, energetico e in altri settori dell'industria moderna.
Tuttavia, alcune caratteristiche dei biocatalizzatori ne rendono inaccettabile l’uso in alcuni casi. Ad esempio, la maggior parte degli enzimi si degrada quando la temperatura aumenta. Gli scienziati stanno cercando di superare tali ostacoli e di aumentare la stabilità degli enzimi in condizioni di produzione difficili utilizzando tecniche di ingegneria proteica.
Oltre alle applicazioni industriali, l’ingegneria proteica ha trovato un posto degno negli sviluppi medici. I ricercatori sintetizzano proteine che possono legarsi e neutralizzare virus e geni mutanti che causano tumori; creando vaccini altamente efficaci e studiando le proteine recettoriali sulla superficie cellulare, che spesso sono bersagli per i prodotti farmaceutici. Gli scienziati alimentari utilizzano l'ingegneria proteica per migliorare le proprietà di conservazione delle proteine vegetali e degli agenti gelificanti o addensanti.
Un altro campo di applicazione dell'ingegneria proteica è la creazione di proteine in grado di neutralizzare sostanze e microrganismi utilizzabili per attacchi chimici e biologici. Ad esempio, gli enzimi idrolasi sono in grado di neutralizzare sia i gas nervini che i pesticidi utilizzati in agricoltura. Inoltre, la produzione, la conservazione e l'utilizzo degli enzimi non sono pericolosi per l'ambiente e la salute umana.
mutagenesi dell'ingegneria proteica modificata
Bibliografia
1. Ingegneria delle proteine.
2. Ingegneria delle proteine. Misteri della genetica. /Vyacheslav Markin // Segreti, enigmi, fatti.
3. Ingegneria delle proteine. // Grande Enciclopedia Russa.
4. Ingegneria delle proteine. // Manuale del chimico 21.
5. Ingegneria proteica ed efficacia dei farmaci.
6. Ingegneria delle proteine. /A.I. Kornelyuk // Biopolimeri e cellule.
7. L’ingegneria proteica migliorerà l’efficacia dei farmaci. // Meccanica popolare.
8. Ingegneria delle proteine. Ricevere insulina. // Biofile - rivista scientifica e di informazione.
9. Biotecnologia. Principali direzioni e risultati. // Biologia per candidati e insegnanti.
10. Bogdanov A.A., Mednikov B.M. Il potere sul gene / A. A. Bogdanov, B. M. Mednikov - M.: Istruzione, 1989 - p.208
11. Ingegneria genetica. // Salute.
12. Geni e chimici. // Genetica.
13. Glick B., Pasternak J. Biotecnologia molecolare. Principi e applicazione / B. Glick, J. Pasternak. - M.: Mir, 2002.
14. Altri ambiti di applicazione dell'ingegneria genetica. /L.V. Timoschenko, M.V. Chubik // Medicina: notizie e tecnologie.
15. Egorova T.A., Klunova S.M., Zhivukhin E.A. Fondamenti di biotecnologia. / T.A. Egorova, S.M. Klunova, E.A. Zhivukhin-M., 2003.
16. Ingegneria delle proteine. // Chimica e biotecnologia.
17. Patrushev L.I. Espressione genica / L.I. Patrushev - M.: Nauka, 2000. - 496 p.
18. Patrushev L.I. Sistemi genetici artificiali. T. 1: Ingegneria genetica e proteica. /L.I. Patrushev - M.: Nauka, 2004. - 526 p.
19. Rybchin V.N. Fondamenti di ingegneria genetica: libro di testo per le università/V.N. Rybchin - San Pietroburgo: Casa editrice dell'Università tecnica statale di San Pietroburgo, 2002. - 522 p.
20. Stepanov V.M. Biologia molecolare. Strutture e funzioni delle proteine. /V.M. Stepanov - M.: Scuola superiore, 1996.
21. Tecnologie biotecnologiche: ingegneria proteica, nanobiotecnologie, biosensori e biochip. / Evgenia Ryabtseva // “Biotecnologia commerciale” - rivista online.
22. Chernavsky D.S., Chernavskaya N.M. Le proteine sono una macchina. Strutture macromolecolari biologiche. /D.S. Chernavsky, N. M. Chernavskaya - M .: Casa editrice dell'Università statale di Mosca, 1999.
23. Schultz G.E., Schirmer R.H. Principi di organizzazione strutturale delle proteine. /G.E. Schultz, R.H. Schirmer-M.: Mir, 1982.
24. Brannigan J.A., Wilkinson A.J. Ingegneria delle proteine 20 anni dopo // Recensioni sulla natura. Biologia cellulare molecolare. 2002.vol. 3. N. 12;
25. Ingegneria delle proteine. // Wikipedia, l'enciclopedia libera.
Pubblicato su Allbest.ru
Documenti simili
L'essenza e i compiti dell'ingegneria genetica, la storia del suo sviluppo. Gli obiettivi della creazione di organismi geneticamente modificati. Inquinamento chimico come conseguenza degli OGM. L'ottenimento dell'insulina umana è il risultato più importante nel campo degli organismi geneticamente modificati.
abstract, aggiunto il 18/04/2013
L'emergere della biotecnologia. Principali direzioni della biotecnologia. La bioenergia come branca delle biotecnologie. Risultati pratici della biotecnologia. Storia dell'ingegneria genetica. Obiettivi, metodi ed enzimi dell'ingegneria genetica. Risultati dell'ingegneria genetica.
abstract, aggiunto il 23/07/2008
Possibilità dell'ingegneria genetica vegetale. Creazione di piante resistenti agli erbicidi. Aumentare l’efficienza della fotosintesi e della fissazione biologica dell’azoto. Migliorare la qualità delle proteine di stoccaggio. Rischi ambientali, medici e socioeconomici dell'ingegneria genetica.
test, aggiunto il 15/12/2011
L'essenza dell'ingegneria genetica, metodi per identificare gli organismi transgenici; produzione e tecnologia degli OGM, differenza rispetto all'allevamento tradizionale, vantaggi e svantaggi. Lo stato e le prospettive per lo sviluppo del mercato dei beni geneticamente modificati nel mondo.
lavoro del corso, aggiunto il 20/11/2010
L'ingegneria genetica è un metodo della biotecnologia che si occupa della ricerca sulla ristrutturazione dei genotipi. Possibilità dell'ingegneria genetica. Prospettive dell'ingegneria genetica. Ridurre il rischio associato alle tecnologie genetiche.
abstract, aggiunto il 04/09/2007
Ingegneria genetica: storia dell'origine, caratteristiche generali, vantaggi e svantaggi. Conoscenza dei più recenti metodi di ingegneria genetica e del loro utilizzo in medicina. Sviluppo dell'ingegneria genetica nel campo dell'allevamento di bestiame e pollame. Esperimenti sui ratti.
lavoro del corso, aggiunto il 07/11/2012
La sequenza delle tecniche di ingegneria genetica utilizzate per creare organismi geneticamente modificati. Classificazione dei principali tipi di enzimi di restrizione utilizzati per la frammentazione del DNA. Enzimi che sintetizzano il DNA su uno stampo di DNA o RNA.
presentazione, aggiunta il 27/04/2014
L'essenza dell'ingegneria genetica e cellulare. I compiti principali della modificazione genetica delle piante, analisi della nocività del loro consumo come cibo. Caratteristiche dell'ibridazione di cellule vegetali e animali. Il meccanismo per ottenere sostanze medicinali mediante l'ingegneria genetica.
presentazione, aggiunta il 26/01/2014
lavoro del corso, aggiunto il 05/10/2011
Fondamenti e tecniche di clonazione del DNA. Fasi dell'ingegneria genetica dei batteri. Sviluppo dell'ingegneria genetica delle piante. Trasformazione genetica e miglioramento delle piante utilizzando agrobatteri, fonti di geni. Sicurezza delle piante geneticamente modificate.
Chimicamente, una proteina è un singolo tipo di molecola, che è una catena di poliamminoacidi o un polimero. È composto da sequenze di aminoacidi di 20 tipi. Dopo aver appreso la struttura delle proteine, le persone si sono poste la domanda: è possibile progettare sequenze di amminoacidi completamente nuove in modo che svolgano le funzioni di cui gli esseri umani hanno bisogno molto meglio delle normali proteine? Il nome migliore per questa idea audace era ingegneria delle proteine.
La gente ha iniziato a pensare a tale ingegneria negli anni '50 del 20 ° secolo. Ciò è avvenuto immediatamente dopo aver decifrato le prime sequenze di aminoacidi proteici. In molti laboratori in tutto il mondo sono stati fatti tentativi per duplicare la natura e sintetizzare chimicamente determinate sequenze di poliamminoacidi assolutamente arbitrarie.
Il chimico B. Merrifield è riuscito maggiormente in questo. Questo americano è riuscito a sviluppare un metodo estremamente efficace per la sintesi delle catene di poliamminoacidi. Per questo alla Merrifield venne assegnato il Premio Nobel per la Chimica nel 1984.
L'americano iniziò a sintetizzare peptidi corti, compresi gli ormoni. Allo stesso tempo, costruì un automa - un "robot chimico" - il cui compito era produrre proteine artificiali. Il robot fece scalpore negli ambienti scientifici. Tuttavia, divenne presto chiaro che i suoi prodotti non potevano competere con ciò che la natura produce.
Il robot non è riuscito a riprodurre accuratamente le sequenze di aminoacidi, ovvero ha commesso degli errori. Ha sintetizzato una catena con una sequenza e un'altra con una leggermente diversa. In una cellula, tutte le molecole di una proteina sono idealmente simili tra loro, cioè le loro sequenze sono assolutamente identiche.
C'era un altro problema. Anche le molecole che il robot ha sintetizzato correttamente non hanno assunto la forma spaziale necessaria per il funzionamento dell'enzima. Pertanto, il tentativo di sostituire la natura con i metodi consueti della chimica organica ha portato a un successo molto modesto.
Gli scienziati potevano solo imparare dalla natura, cercando le necessarie modifiche delle proteine. Il punto è che in natura avvengono costantemente mutazioni che portano a cambiamenti nelle sequenze di aminoacidi delle proteine.
Se selezioni mutanti con le proprietà necessarie, ad esempio quelli che elaborano un particolare substrato in modo più efficiente, puoi isolare da tale mutante un enzima alterato, grazie al quale la cellula acquisisce nuove proprietà. Ma questo processo richiede un periodo di tempo molto lungo.
Tutto è cambiato quando è apparsa l’ingegneria genetica. Grazie a lei, hanno iniziato a creare geni artificiali con qualsiasi sequenza nucleotidica. Questi geni sono stati inseriti in molecole vettoriali preparate e il DNA è stato introdotto in batteri o lieviti. Lì, una copia dell'RNA è stata prelevata dal gene artificiale. Di conseguenza, è stata prodotta la proteina richiesta. Sono stati esclusi errori nella sua sintesi. La cosa principale era selezionare la giusta sequenza di DNA, dopodiché il sistema enzimatico della cellula avrebbe svolto il suo lavoro in modo impeccabile.
Possiamo quindi concludere che l’ingegneria genetica ha aperto la strada all’ingegneria proteica nella sua forma più radicale. Ad esempio, abbiamo scelto una proteina e volevamo sostituire un residuo di amminoacido in essa contenuto con un altro.
Prima di iniziare il lavoro di sostituzione, è necessario preparare un vettore del DNA. Si tratta di DNA virale o plasmidico in cui è incorporato il gene della proteina che ci interessa. È inoltre necessario conoscere la sequenza nucleotidica del gene e la sequenza aminoacidica della proteina codificata. Quest'ultimo viene determinato dal primo utilizzando una tabella di codici genetici.
Utilizzando la tabella, è anche facile determinare quali modifiche minime dovrebbero essere apportate alla composizione del gene in modo che inizi a codificare non l'originale, ma una proteina modificata su nostra richiesta. Diciamo che nel mezzo di un gene bisogna sostituire la guanina con la timina.
A causa di una cosa così piccola, non è necessario risintetizzare l'intero gene. Viene sintetizzato solo un piccolo frammento di nucleotidi, complementare alla regione al centro della quale si trova il nucleotide guanina scelto per la sostituzione.
Il frammento risultante viene mescolato con un vettore di DNA (DNA circolare), che contiene il gene di cui abbiamo bisogno. L'anello del DNA e il frammento sintetizzato creano una sezione della doppia elica Watson-Crick. In esso, la coppia centrale viene “espulsa” dalla doppia elica, poiché è formata da nucleotidi reciprocamente non complementari.
Aggiungere quattro dNTP e DNA polimerasi alla soluzione. Quest'ultimo, utilizzando un frammento attaccato ad un unico anello, lo completa fino ad ottenere un anello completo nel pieno rispetto del principio di complementarità.
Di conseguenza, otteniamo un DNA vettoriale quasi normale. Può essere introdotto in un lievito o in una cellula batterica per la riproduzione. L'unica cosa è che questo DNA differisce dal vettore originale in una coppia non complementare. In altre parole, l’elica vettoriale del DNA non è del tutto perfetta.
Al primissimo atto di raddoppiare il vettore risultante insieme ai batteri che lo trasportano, ciascuna delle molecole di DNA figlia diventerà una doppia elica perfetta su tutta la sua lunghezza. Tuttavia, una delle molecole figlie porta la coppia di nucleotidi originali e l'altra ha un vettore mutante in questo posto, sulla base del quale si ottiene la proteina mutante che ci interessa.
Pertanto, l’ingegneria proteica crea una miscela di cellule. Alcune di esse portano il vettore originale con un gene non mutante, mentre altre cellule portano il gene mutante. Resta da selezionare da questa miscela esattamente quelle cellule in cui si trova il gene mutante.
I metodi di ingegneria genetica, in particolare la clonazione di singoli geni o parti di essi, nonché il sequenziamento del DNA, hanno permesso di migliorare significativamente la metodologia della mutagenesi, eliminando i principali svantaggi dei metodi classici per indurre mutazioni nei genomi. L'analisi genetica classica prevede l'effetto di un fattore mutageno in vivo sull'intero genoma, a seguito del quale si verificano mutazioni casuali, spesso multiple, che complicano notevolmente l'identificazione dei mutanti. Gli individui mutanti vengono identificati dalle caratteristiche fenotipiche alterate e la natura della mutazione può essere determinata dopo il sequenziamento del DNA. La moderna mutagenesi localizzata, infatti, prevede azioni inverse: prima viene clonato il gene di interesse o il suo segmento, la sua struttura viene determinata durante il sequenziamento, quindi vengono apportate le modifiche necessarie alla sua composizione in vitro. Le conseguenze della mutazione indotta vengono determinate dopo l'introduzione del gene mutante nell'organismo originale.
La versione più semplice della mutagenesi localizzata consiste nel trattare un frammento di DNA clonato con uno dei fattori mutageni, ma il risultato di tale esposizione sarà anche un cambiamento casuale nella struttura del frammento. Metodi più affidabili e più spesso utilizzati di mutagenesi localizzata vengono eseguiti senza l'uso di fattori mutageni. Tra i tipi di mutazioni predominano le delezioni, le inserzioni e le sostituzioni nucleotidiche.
Eliminazioni. Questi tipi di mutazioni nella mutagenesi localizzata sono ottenuti utilizzando endonucleasi. Vengono utilizzate sia endonucleasi di restrizione che non specifiche. Il caso più semplice di utilizzo degli enzimi di restrizione è quello di scindere un genoma utilizzando un enzima di restrizione che introduce diverse rotture per formare estremità appiccicose. I frammenti risultanti vengono nuovamente chiusi in un anello utilizzando la DNA ligasi, che può portare alla formazione di molecole che non contengono uno dei segmenti di DNA. Questo approccio produce delezioni estese e viene tipicamente utilizzato in esperimenti preliminari per determinare la funzione di sezioni relativamente grandi di DNA clonato.
Piccole delezioni si ottengono come segue. Il frammento clonato viene tagliato all'interno del vettore nel sito appropriato utilizzando un enzima di restrizione (Fig. 21.1). La molecola lineare risultante viene trattata con esonucleasi III, che idrolizza un filamento di DNA,
a partire dalla fine dei 3'. Il risultato è un insieme di molecole con code da 5' a filamento singolo di varia lunghezza. Queste code vengono idrolizzate dalla nucleasi S1 specifica per ssDNA e si formano delezioni nel DNA. È inoltre possibile utilizzare l'esonucleasi Bal 31, che catalizza la degradazione di entrambi i filamenti, a partire dalle estremità delle molecole lineari di DNA. Il decorso delle reazioni nucleotiche è regolato variando il tempo di incubazione, la temperatura e la concentrazione dell'enzima, inducendo la formazione di delezioni di diversa lunghezza. Le varianti di delezione risultanti del DNA lineare sono spesso dotate di linker prima della ciclizzazione in modo che nella regione della delezione siano presenti siti di restrizione. Ci sono altre modifiche ai metodi descritti.
Inserzioni (inserimenti). Per ottenere le inserzioni, il DNA clonato viene digerito con un enzima di restrizione o endonucleasi aspecifica, quindi i frammenti risultanti vengono legati in presenza del segmento che si vuole inserire nel DNA. Molto spesso, come tali segmenti vengono utilizzati i polylinker sintetizzati chimicamente (capitolo 20).
Le inserzioni, come le delezioni, possono distruggere l'integrità di un gene o la struttura delle sue regioni regolatrici, determinando la sintesi di una proteina difettosa (nel caso di delezioni estese o frameshift, solitamente inattiva) o cambiamenti nel processo di trascrizione del gene di interesse. In questo modo si ottengono spesso mutanti regolatori e si costruiscono vettori di espressione (Capitolo 20).
Mutazioni puntiformi . Queste mutazioni sono sostituzioni nucleotidiche. Per ottenerli si possono utilizzare diversi approcci: deaminazione della citosina, incorporazione di analoghi nucleotidici, incorporazione errata di nucleotidi durante la riparazione dei gap, ecc.
Il primo metodo si basa sul fatto che i residui di citosina nel DNA a filamento singolo possono essere deaminati per formare uracile mediante trattamento con ioni bisolfito. Le regioni a filamento singolo nel DNA vengono solitamente ottenute vicino ai siti di restrizione, ad esempio mediante l'azione dell'esonucleasi III. Dopo il trattamento con bisolfito, gli spazi vuoti a filamento singolo vengono riempiti utilizzando la DNA polimerasi e le estremità vengono legate. Nei siti in cui si è formato l'uridilato invece del citidilato durante la deaminazione, l'adenilato occuperà la posizione complementare e durante la replicazione di tale molecola la coppia GC sarà sostituita da una coppia AT.
Un altro approccio per indurre sostituzioni consiste nel trattare il DNA clonato con un enzima di restrizione in presenza di bromuro di etidio, che si inserisce tra i piani delle coppie di basi e distrugge la struttura del duplex. Di conseguenza, si forma solo una rottura del DNA a filamento singolo. Viene creato un piccolo spazio nel sito della rottura del singolo filamento e quindi riparato in presenza di DNA polimerasi, dATP, dGTP, dCTP e N-4-idrossicitosina trifosfato invece di dTTP. L'idrossicitosina trifosfato è inclusa nella catena al posto del timidilato, ma durante la replicazione del DNA si accoppia ugualmente bene sia con l'adenilato che con il guanilato. Come risultato dell'inclusione del guanilato dopo un ulteriore ciclo di replicazione, la sostituzione AT→GC avverrà in questo sito (Fig. 21.2). Poiché in questo metodo la sostituzione dei nucleotidi viene effettuata internamente
sito di restrizione, diventa possibile distinguere facilmente tra vettori con la sequenza originale e vettori mutanti. Per fare ciò è sufficiente trattarle con l’enzima di restrizione utilizzato nell’esperimento: le molecole mutanti non subiranno il clivaggio.
Un metodo simile si basa sull'utilizzo di solo tre dei quattro possibili nucleotidi quando si riempie un gap a filamento singolo con la DNA polimerasi. Nella maggior parte dei casi, l'enzima si ferma nel punto della molecola in cui si trova il nucleotide complementare a quello mancante. Tuttavia, occasionalmente, la DNA polimerasi commette un errore e attiva uno dei tre nucleotidi presenti. Ciò porta alla formazione di molecole ad anello, che contengono basi azotate non complementari spaiate. Quando tali vettori vengono introdotti nelle cellule batteriche, alcune molecole subiranno la riparazione di tale danno. Di conseguenza, in metà delle molecole dopo la replicazione verrà ripristinata la sequenza originale e nell'altra metà la mutazione verrà fissata. Le molecole mutanti possono essere distinte utilizzando il metodo sopra descritto.
Mutagenesi sito-specifica. I metodi caratterizzati di mutagenesi localizzata differiscono in quanto i siti in cui si verificano le mutazioni vengono selezionati in modo casuale. Allo stesso tempo, la tecnica della mutagenesi sito-specifica rende possibile introdurre mutazioni in una regione del gene precisamente definita. Questo viene fatto utilizzando oligonucleotidi sintetici (ottenuti mediante sintesi chimica) con una determinata sequenza. Il metodo è conveniente in quanto non richiede la presenza di convenienti siti di restrizione. Il metodo si basa sulla formazione di eteroduplex tra un oligonucleotide sintetico contenente una mutazione e un DNA complementare a filamento singolo nel vettore.
Procedi come segue. Viene sintetizzato un piccolo oligonucleotide (8-20 monomeri), complementare alla parte del gene in cui si vuole ottenere una mutazione. Sono consentite una o più sostituzioni nucleotidiche nella regione centrale dell'oligonucleotide. Il gene in studio o il suo frammento viene clonato come parte di un vettore basato sul fago M13 per ottenere DNA ricombinante circolare a filamento singolo. I vettori ricombinanti vengono miscelati e ricotti con oligonucleotidi. Si verifica l'ibridazione dell'oligonucleotide con la regione complementare, mentre i nucleotidi non complementari rimangono spaiati. L'oligonucleotide agisce come primer in una reazione della polimerasi che coinvolge la DNA polimerasi in vitro. L'anello è chiuso da ligasi. La molecola circolare risultante viene introdotta nelle cellule di E. coli, dove avviene la riparazione parziale dei siti di replicazione mutante. La frequenza delle mutazioni varia solitamente dall'1 al 50%. La selezione delle cellule contenenti molecole di DNA mutanti può essere effettuata in diversi modi, tra i quali il vantaggio è il metodo che utilizza un oligonucleotide marcato radioattivamente, che viene utilizzato per la mutagenesi. In questo caso, questo nucleotide funge da sonda. Il principio di utilizzo di tale sonda si basa sul fatto che è completamente complementare al DNA mutante e parzialmente complementare al DNA wild-type. È possibile selezionare condizioni di ibridazione tali (principalmente temperatura) che l'ibridazione della sonda marcata sarà stabile solo con la sequenza di DNA mutante, che può essere rilevata mediante autoradiografia.
Il metodo della mutagenesi sito-specifica è particolarmente prezioso perché consente di isolare le mutazioni senza controllarne la manifestazione fenotipica. Questo metodo apre nuove opportunità per studiare le funzioni degli elementi regolatori dei geni, consente di modificare la "forza" dei promotori, ottimizzare i siti di legame dei ribosomi, ecc. Una delle principali applicazioni di questa metodologia è ingegneria delle proteine.
Ingegneria delle proteine. Con questa frase si indica un insieme di tecniche metodologiche che rendono possibile ricostruire una molecola proteica mediante l'introduzione mirata di mutazioni appropriate in un gene strutturale (mutagenesi sito-specifica) e, di conseguenza, le sostituzioni di amminoacidi desiderate nella struttura primaria della proteina.
Un esempio illustrativo della costruzione di proteine più attive sono gli esperimenti di Fersht e collaboratori con l'enzima tirosil-tRNA sintetasi del batterio Bacillus stearothermophilus. L'analisi delle conseguenze delle sostituzioni di amminoacidi nel sito attivo di questo enzima ha portato alla conclusione che la rimozione dei gruppi che formano deboli legami idrogeno con il substrato può migliorare la sua affinità per il substrato. Si è scoperto che la treonina-51 (occupa la posizione 51 nel peptide) forma un legame idrogeno lungo e debole con l'ossigeno dell'anello di ribosio quando lega il tirosil adenilato. Allo stesso tempo, si è scoperto che la prolina occupa la stessa posizione nei batteri E. coli. La mutagenesi sito-specifica del gene che determina la struttura della tirosil-tRNA sintetasi di B.stearothermophilus ha reso possibile la sostituzione thr-51→pro -51 nel peptide. Di conseguenza, il legame dell'ATP nel centro attivo dell'enzima è migliorato notevolmente e la sua attività catalitica è aumentata di 25 volte.
Un altro esempio non meno significativo di ricostruzione proteica di importanza pratica è la modifica della subtilisina da Bacillus amyloliquefaciens, effettuata da Estell et al. Le subtilisine sono proteinasi seriniche secrete dai bacilli nell'ambiente esterno. Questi enzimi sono prodotti su larga scala dall'industria biotecnologica e sono ampiamente utilizzati nei detergenti. Lo svantaggio delle subtilisine è una forte diminuzione dell'attività proteolitica sotto l'influenza di agenti ossidanti, compresi quelli contenuti nei detersivi. L'obiettivo della ricostruzione della molecola di subtilisina BPN era stabilizzarla contro l'ossidazione chimica.
In esperimenti preliminari si è riscontrato che in presenza di acqua ossigenata la subtilisina riduce rapidamente l'attività a causa dell'ossidazione del residuo di metionina-222, che viene convertito nel corrispondente solfossido. Utilizzando metodi di mutagenesi sito-specifici, questo residuo di metionina è stato sostituito con tutti gli altri 19 aminoacidi proteici. Plasmidi con geni mutanti sono stati introdotti in ceppi con delezioni nei geni corrispondenti e sono state analizzate le proprietà delle subtilisine prodotte. I mutanti con serina e alanina222 si sono rivelati abbastanza stabili all'azione del perossido. Il mutante più attivo è stato quello contenente il residuo di cisteina-222; la sua attività specifica era superiore del 38% rispetto a quella del ceppo wild-type.
In modo simile è stato possibile aumentare l'attività dell'interferone-β. Altri risultati dell'ingegneria proteica includono studi volti a chiarire l'attività di trasformazione delle oncoproteine; modificare la termostabilità degli enzimi, ad esempio, ottenendo renina termolabile e a-amilasi termostabile; aumento dell'efficienza del legame dell'insulina da parte del corrispondente recettore della membrana plasmatica grazie alla sostituzione dell'istidina con aspartato nella posizione 10 della catena b dell'ormone, così come molti altri esempi. Un gran numero di prodotti di ingegneria proteica hanno già trovato applicazione pratica nei processi di produzione.
1.1 Il concetto di ingegneria proteica. Storia dello sviluppo
L'ingegneria proteica è una branca della biotecnologia che si occupa dello sviluppo di proteine utili o preziose. Si tratta di una disciplina relativamente nuova che si concentra sullo studio del ripiegamento delle proteine e sui principi della modifica e creazione delle proteine.
Esistono due strategie principali per l'ingegneria proteica: la modificazione diretta delle proteine e l'evoluzione diretta. Questi metodi non si escludono a vicenda; i ricercatori spesso usano entrambi. In futuro, una conoscenza più dettagliata della struttura e della funzione delle proteine, nonché i progressi nell'alta tecnologia, potrebbero espandere significativamente le possibilità dell'ingegneria proteica. Di conseguenza, anche gli aminoacidi innaturali possono essere incorporati grazie a un nuovo metodo che consente di incorporare nuovi aminoacidi nel codice genetico.
L'ingegneria delle proteine ha avuto origine all'intersezione tra la fisica e la chimica delle proteine e l'ingegneria genetica. Risolve il problema della creazione di molecole proteiche modificate o ibride con caratteristiche specifiche. Un modo naturale per eseguire tale compito è prevedere la struttura del gene che codifica per la proteina alterata, effettuarne la sintesi, la clonazione e l'espressione nelle cellule riceventi.
La prima modificazione controllata delle proteine fu effettuata a metà degli anni '60 da Koshland e Bender. Per sostituire il gruppo idrossile con un gruppo sulfidrile nel centro attivo della proteasi, la subtilisina, hanno utilizzato un metodo di modificazione chimica. Tuttavia, come si è scoperto, tale tiolsubtilisina non mantiene l'attività della proteasi.
Chimicamente, una proteina è un singolo tipo di molecola, che è una catena di poliamminoacidi o un polimero. È composto da sequenze di aminoacidi di 20 tipi. Dopo aver appreso la struttura delle proteine, le persone si sono poste la domanda: è possibile progettare sequenze di amminoacidi completamente nuove in modo che svolgano le funzioni di cui gli esseri umani hanno bisogno molto meglio delle normali proteine? Il nome Protein Engineering era appropriato per questa idea.
La gente ha iniziato a pensare a tale ingegneria negli anni '50 del 20 ° secolo. Ciò è avvenuto immediatamente dopo aver decifrato le prime sequenze di aminoacidi proteici. In molti laboratori in tutto il mondo sono stati fatti tentativi per duplicare la natura e sintetizzare chimicamente determinate sequenze di poliamminoacidi assolutamente arbitrarie.
Il chimico B. Merrifield è riuscito maggiormente in questo. Questo americano è riuscito a sviluppare un metodo estremamente efficace per la sintesi delle catene di poliamminoacidi. Per questo alla Merrifield venne assegnato il Premio Nobel per la Chimica nel 1984.
Figura 1. Schema di come funziona l'ingegneria proteica.
L'americano iniziò a sintetizzare peptidi corti, compresi gli ormoni. Allo stesso tempo, costruì un automa - un "robot chimico" - il cui compito era produrre proteine artificiali. Il robot fece scalpore negli ambienti scientifici. Tuttavia, divenne presto chiaro che i suoi prodotti non potevano competere con ciò che la natura produce.
Il robot non è riuscito a riprodurre accuratamente le sequenze di aminoacidi, ovvero ha commesso degli errori. Ha sintetizzato una catena con una sequenza e l'altra con una leggermente modificata. In una cellula, tutte le molecole di una proteina sono idealmente simili tra loro, cioè le loro sequenze sono assolutamente identiche.
C'era un altro problema. Anche le molecole che il robot ha sintetizzato correttamente non hanno assunto la forma spaziale necessaria per il funzionamento dell'enzima. Pertanto, il tentativo di sostituire la natura con i metodi consueti della chimica organica ha portato a un successo molto modesto.
Gli scienziati potevano solo imparare dalla natura, cercando le necessarie modifiche delle proteine. Il punto è che in natura avvengono costantemente mutazioni che portano a cambiamenti nelle sequenze di aminoacidi delle proteine. Se selezioni mutanti con le proprietà necessarie che elaborano un particolare substrato in modo più efficiente, puoi isolare da tale mutante un enzima alterato, grazie al quale la cellula acquisisce nuove proprietà. Ma questo processo richiede un periodo di tempo molto lungo.
Tutto è cambiato quando è apparsa l’ingegneria genetica. Grazie a lei, hanno iniziato a creare geni artificiali con qualsiasi sequenza nucleotidica. Questi geni sono stati inseriti in molecole vettoriali preparate e il DNA è stato introdotto in batteri o lieviti. Lì, una copia dell'RNA è stata prelevata dal gene artificiale. Di conseguenza, è stata prodotta la proteina richiesta. Sono stati esclusi errori nella sua sintesi. La cosa principale era selezionare la giusta sequenza di DNA, dopodiché il sistema enzimatico della cellula avrebbe svolto il suo lavoro in modo impeccabile. Possiamo quindi concludere che l’ingegneria genetica ha aperto la strada all’ingegneria proteica nella sua forma più radicale.
Ingegneria delle proteine
Modificazione proteica mirata. Nella modifica mirata delle proteine, lo scienziato utilizza la conoscenza dettagliata della struttura e della funzione della proteina per apportare le modifiche desiderate. In generale, questo metodo ha il vantaggio...
Ingegneria delle proteine
La tecnologia dell'ingegneria proteica viene utilizzata (spesso in combinazione con il metodo del DNA ricombinante) per migliorare le proprietà delle proteine esistenti (enzimi, anticorpi, recettori cellulari) e creare nuove proteine che non esistono in natura...
Ingegneria delle proteine
1. Sostituendo diversi residui amminoacidici del lisozima del batteriofago T4 con cisteina, è stato ottenuto un enzima con un gran numero di legami disolfuro, grazie al quale questo enzima ha mantenuto la sua attività a una temperatura più elevata. 2...
Specie e speciazione
Aristotele usava il termine "specie" per descrivere animali simili. Dopo la comparsa delle opere di D. Ray (1686) e soprattutto di C. Linnaeus (1751-- 1762), il concetto di specie si è saldamente affermato in biologia come il principale...
Maggiore attività nervosa in età adulta
Il funzionamento del cervello è rimasto per molti anni un mistero irrisolto per l’umanità. Non solo il clero, ma anche gli scienziati che professavano l'idealismo collegavano tutti i processi mentali del corpo con un'anima misteriosa...
Algoritmi genetici nel problema dell'ottimizzazione di parametri reali
Quello che viene chiamato algoritmo genetico standard è stato descritto ed esplorato in dettaglio per la prima volta nel lavoro di de Jongh...
Ingegneria genetica
L'ingegneria genetica è apparsa grazie al lavoro di numerosi ricercatori in vari rami della biochimica e della genetica molecolare. Per molti anni le proteine sono state considerate la principale classe di macromolecole. C'era anche un'ipotesi...
L'uso dell'ingegneria genetica nella cura delle malattie e nella creazione di medicinali
L'ingegneria genetica è apparsa grazie al lavoro di numerosi ricercatori in vari rami della biochimica e della genetica molecolare...
Storia della genetica
Dopo la capillare diffusione degli insegnamenti di Charles Darwin, uno dei primi critici a evidenziare un punto debole della teoria fu il ricercatore scozzese F. Jenkins. Nel 1867 notò che nella teoria di Darwin non c'era chiarezza sulla questione...
Concetti per lo sviluppo delle moderne tecnologie ed energia
Per facilitare il lavoro fisico, fin dai tempi antichi sono stati inventati vari dispositivi, meccanismi e macchine che migliorano le capacità meccaniche umane. Ma solo pochi meccanismi aiutavano una persona a svolgere il lavoro...
Caratteristiche della clonazione
Razze di polli e loro distribuzione moderna
L'allevamento di pollame nella maggior parte dei paesi del mondo occupa una posizione di leadership tra gli altri rami della produzione agricola, fornendo alla popolazione prodotti alimentari dietetici di grande valore (uova, carne, delizioso fegato grasso)...
Il problema dell’esistenza dell’umanità alla luce della teoria della noosfera di Vernadsky
Basandosi sull'osservazione dei fenomeni naturali, l'idea che gli esseri viventi interagiscono con l'ambiente esterno e ne influenzano i cambiamenti è nata molto tempo fa...
La citogenetica come scienza
La citogenetica è la scienza delle basi materiali dell'ereditarietà. Studia le caratteristiche della struttura, riproduzione, ricombinazione, cambiamenti e funzionamento delle strutture genetiche della cellula, la loro distribuzione nella mitosi...
Evoluzione di gruppi di organismi
La teoria evoluzionistica è la dottrina dei modelli generali e delle forze trainanti dello sviluppo storico della natura vivente. Lo scopo di questo insegnamento: identificare modelli di sviluppo del mondo organico per la successiva gestione di questo processo...

Ingegneria delle proteine 6 Una serie di metodi e approcci per studiare le proteine e ottenere proteine con nuove proprietà COMPITI PRINCIPALI Creare una libreria di cloni di sequenze nucleotidiche e di amminoacidi Studiare gli effetti di singole sostituzioni di residui di amminoacidi sul ripiegamento e sulle funzioni delle proteine Sviluppare metodi per modificare efficacemente proteine per conferire loro le proprietà necessarie Sviluppare metodi e approcci per lo screening e la selezione delle proteine con le proprietà richieste


Progettazione razionale Progettazione razionale La necessità di conoscere l'organizzazione spaziale della proteina La necessità di conoscere le interazioni intra e intermolecolari L'imperfezione dei metodi e delle attrezzature una direzione volta a creare nuove proteine de novo attraverso la loro progettazione spaziale

L'evoluzione diretta delle molecole proteiche è una direzione volta a creare nuove proteine attraverso la selezione 1 ottenendo una libreria di sequenze di amminoacidi casuali 2 selezionando catene polipeptidiche che abbiano almeno un piccolo grado delle proprietà richieste 3 utilizzando la mutagenesi casuale ottenendo una nuova libreria di proteine che sono utilizzati nel successivo ciclo di selezione o utilizzando costrutti geneticamente modificati che esprimono nuove proteine

Evoluzione diretta delle molecole proteiche (opzioni) riprogettazione razionale mediante mutagenesi diretta sostituzione di specifici residui di amminoacidi nel centro attivo dell'ingegneria enzimatica delle superfici proteiche mediante mutazioni modifica di sezioni della catena polipeptidica in prossimità di residui di amminoacidi che sono vicini tra loro sulla superficie superficie del globulo proteico, ma situati a notevole distanza l'uno dall'altro nella catena polipeptidica

Screening e selezione di proteine con proprietà specifiche screening casuale selezione di screening migliorata ogni proteina viene esaminata per verificare la presenza delle proprietà richieste; la selezione delle proteine dalla libreria avviene in modo casuale; ogni proteina viene esaminata per verificare la presenza delle proprietà richieste; la selezione delle proteine dalla libreria avviene in modo casuale; è possibile se gli oggetti che compongono la libreria differiscono fenotipicamente (ad esempio, in presenza di attività enzimatica); si creano le condizioni per la conservazione selettiva dei componenti della libreria che hanno determinate proprietà (phage, cell display); vengono create le condizioni per la conservazione selettiva dei componenti della libreria; che hanno determinate proprietà (phage, cell display) rilevamento di una proteina con le proprietà richieste tra un gran numero di macromolecole che compongono la libreria clone risultante


Visualizzazione dei fagi L'obiettivo è visualizzare le proteine estranee sulla superficie del fago. Il metodo è stato sviluppato nel 1985 per il batteriofago filamentoso M13. (i geni pIII e pVIII sono siti bersaglio adatti per l'inserimento di un frammento di cDNA estraneo) L'obiettivo è esporre proteine estranee sulla superficie del fago.Il metodo è stato sviluppato nel 1985 per il batteriofago filamentoso M13. (i geni pIII e pVIII sono siti bersaglio adatti per l'inserimento di un frammento di cDNA estraneo) viene costruito un gene ibrido costituito dalle sequenze codificanti della proteina bersaglio e da una delle proteine dell'involucro fagico da parte del batteriofago; E. coli viene infettato durante la fagizzazione assemblaggio; le proteine ibride sono incluse nella particella fagica

Fago fago helper Genoma fagico Infezione di E.coli con un fago helper Le cellule di E.coli trasformate con una libreria plasmidica/fagemide vengono infettate con un fago helper per ottenere particelle fagiche sulla cui superficie sono esposte varie varianti della proteina bersaglio E. coli trasformate con una libreria plasmidica/fagemide, vengono infettate con un fago helper per ottenere particelle fagiche sulla cui superficie sono esposte varie varianti della proteina bersaglio


Prospettive per l'uso pratico dell'ingegneria proteica Medicina: *per la produzione di nuovi farmaci; per la creazione di strumenti diagnostici e la produzione di vaccini; *per studiare i meccanismi della risposta immunitaria, nonché le malattie del sistema immunitario Ecologia: *per ottenere biocatalizzatori sotto forma di cellule intere con enzimi immobilizzati sulla loro superficie; *per l'ottenimento di biosensori per la diagnostica e il monitoraggio ambientale; *per la creazione di bioadsorbenti per rimuovere sostanze tossiche e ioni di metalli pesanti dall'ambiente

Misurazione del glucosio utilizzando un elettrodo enzimatico (rappresentazione schematica dell'esperimento di L. Clark). Ossidazione del glucosio da parte dell'enzima glucosio ossidasi in presenza di ossigeno: glucosio + O 2 H 2 O 2 + glucono-1,5-lattone. Sull'elettrodo di platino viene ridotta H 2 O 2 ad un potenziale di +700 mV; la corrente che circola nel circuito è proporzionale alla concentrazione di acqua ossigenata (cioè, indirettamente, glucosio).